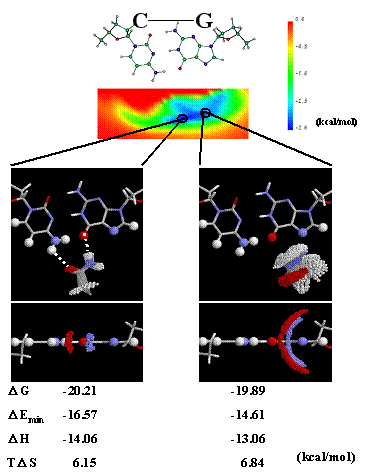
図1.アスパラギン側鎖の自由エネルギーマップ(上)および有利な領域における代表的な構造(下)
DNAと蛋白質との特異的相互作用

図1.アスパラギン側鎖の自由エネルギーマップ(上)および有利な領域における代表的な構造(下)
遺伝子の発現とその制御には,ある塩基配列を特異的に認識し結合する蛋白質が重要な役割を果たしていると考えられている。しかし,その特異的な認識がどのようなメカニズムでおこっているのかについてはわかっていない。その機構を明らかにする目的で,単に相互作用のポテンシャルエネルギーを求めるだけではなく,側鎖が様々な構造をとりうることも考慮にいれた自由エネルギーマップという概念を提案した。実際にマップを計算することによって蛋白質側鎖と核酸塩基対の特異的な認識はこの自由エネルギーマップから理解できることを示した[1,2]。
塩基対のmajor groove側で塩基対の平面上にメッシュを切り,その格子点上にアミノ酸側鎖のCa原子をおく。その点にCa原子がある場合に,側鎖の構造を系統的に発生させることによってすべての構造を網羅し,それらのエネルギーを計算する。アスパラギン側鎖の場合,一つの格子点で約230万点の構造を発生させる。格子点の数は約500なので,一つのマップには約10億個の構造の情報が含まれていることになる。図1には,C−G塩基対とアスパラギン側鎖間の自由エネルギーマップを示す。
統計処理を施すためには非常に多数の構造を発生させ,それらに対するエネルギー計算を実行する必要がある。しかし,10億個のオーダーのab initio MO法計算を実行することは事実上不可能である。そこで,ab initio MO法計算を再現するポテンシャルセットをあらかじめ作成し,そのポテンシャル関数を使うことによって,ab initio MO法計算をした場合と同じ結果であると期待できる計算結果を得る,という手法を用いる。いくら長いシミュレーションを実行してもそこで使ったポテンシャルが信頼できないのならばその結果は信用できない,ということは直視すべき真理である。ここでの私達の大量計算は,核酸と蛋白質間の相互作用のためにab initio MO法計算の結果をフィッティングすることによって作られた ab initio potential [3] を使用して実行している。
図1(上)のマップにおいて,青い領域が,アスパラギン側鎖とC−G塩基対との相互作用が自由エネルギー的に有利な領域である。図1(下)には,同じように有利な領域上の2点について,関与する構造を示した。図1(下)において,分布している赤い点はアスパラギン側鎖のカルボニル基の酸素原子,青い点はアミノ基の窒素原子,白い点は水素原子である。図1(下)の左図と右図とを比較すると,左側の方がポテンシャルエネルギー的にはかなり安定な構造である。しかし右図の方が同じような安定性を示す構造が幅広く分布している。したがって自由エネルギー的には同じような有利さを示している。
このように,自由エネルギーマップを描くことにより,単に最も安定な構造を得ることから得られるものとは異なる描像があらわれてくる。生体系のようにさまざまな要因が複雑にからみあいながら,全体としてある方向に反応がすすんでいくような系を理解するためには,このように「同じようなポテンシャルエネルギーをもつ多くの場合がありうる」ということをふまえた解析が必要なのである。
原著論文:
[1] Fabio Pichierre, Misako Aida, M. Michael Gromiha and Akinori Sarai, "Free-energy maps of base-amino acid interactions for DNA-protein recognition," Journal of the American Chemical Society, 121, 6152-6157 (1999).
[2] Tomoki Yoshida, Tomohiro Nishimura, Misako Aida1, Fabio Pichierri, Michael M. Gromiha and Akinori Sarai, "Evaluation of free energy landscape for base-amino acid interactions using ab initio force field and extensive sampling," Biopolymers, 61, 84-95 (2002).
[3] Misako Aida, Giorgina Corongiu and Enrico Clementi, "Ab initio force field for simulations of proteins and nucleic acids," International Journal of Quantum Chemistry, 42, 1353-1381 (1992).